Cell Metab:多组学揭示2型糖尿病的遗传因素和环境压力作用
来源:生物探索 2024-10-25 11:42
这项研究将遗传突变和环境因素的变化与胰岛细胞的转录影响关联起来,为理解T2D发病机制以及开发潜在的治疗方法提供了重要的借鉴意义。
2型糖尿病(T2D)是一种常见的慢性代谢性疾病,其特点是身体不能有效利用胰岛素(胰岛素抵抗)或胰腺无法分泌足够的胰岛素,导致血糖水平升高【1-3】。胰岛素是一种由胰腺β细胞分泌的激素,帮助细胞摄取和利用血糖。当身体对胰岛素不敏感时,胰腺必须分泌更多的胰岛素来维持正常的血糖水平。随着时间的推移,胰腺可能无法跟上这种需求,导致胰岛素不足,进而引发高血糖。
T2D的主要风险因素包括不健康的饮食、缺乏运动、超重或肥胖、遗传因素和年龄的增长。该疾病通常起病缓慢,可能无明显症状,但长期高血糖可引发心脏病、中风、肾病、视力丧失等严重并发症。由于遗传和环境因素共同作用导致T2D的发病,因此GWASs研究已经将超过600个序列突变与T2D关联起来【4,5】。这些变异大多位于非编码区,与胰腺β细胞的顺式调控元件(CREs)高度富集有关,表明这些变异可能通过改变CRE功能和效应基因表达,导致胰岛β细胞功能障碍和衰竭。T2D的发病过程涉及遗传变异与环境应激因素(如内质网(ER)应激和炎性细胞因子应激)的动态交互作用。然而,目前关于非编码区变异如何在这些应激条件下影响胰岛的转录反应,了解仍十分有限。
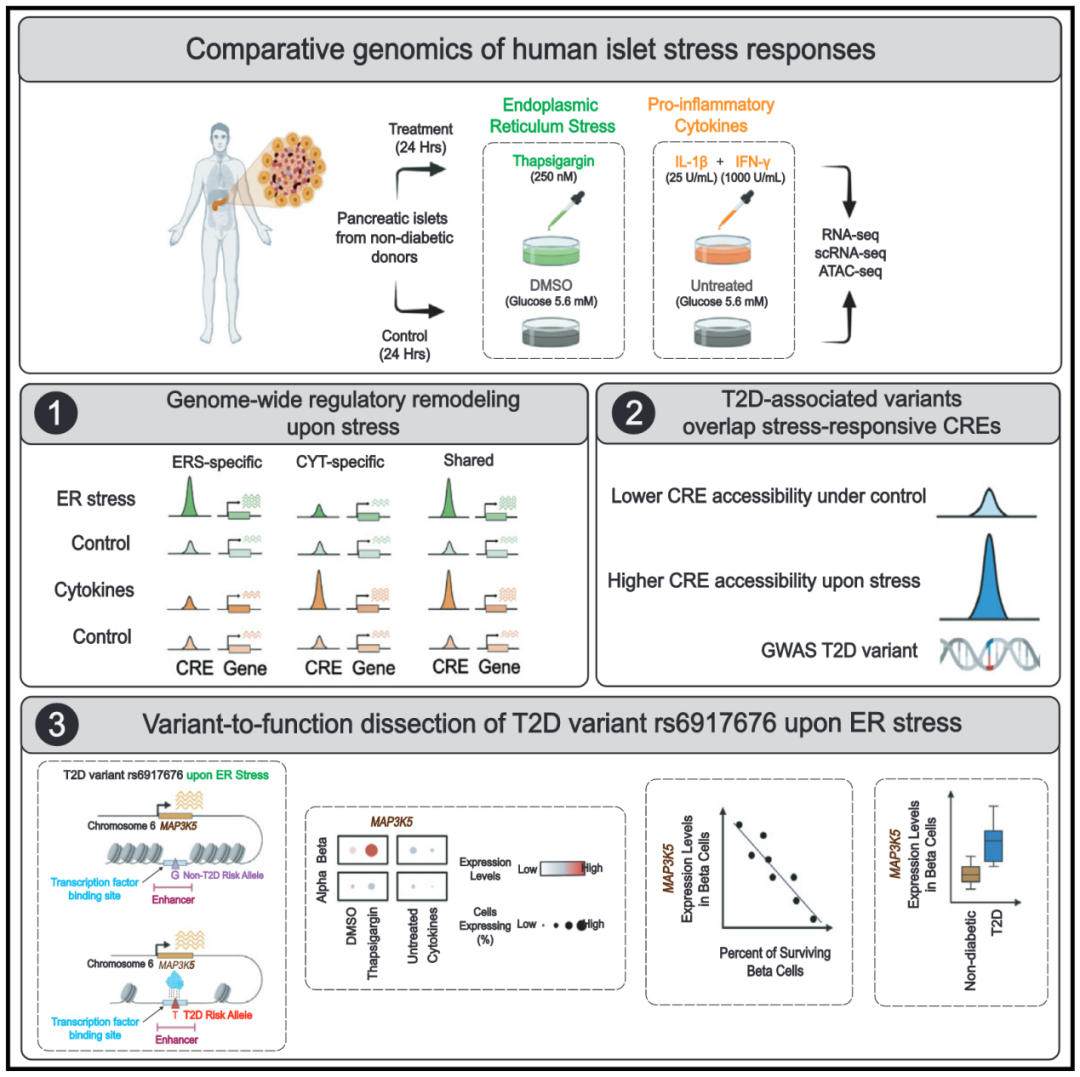
近日,来自美国杰克逊基因组医学实验室的Michael L. Stitzel和Duygu Ucar合作在Cell Metabolism上发表了Resource类论文Multi-omic mapping of human pancreatic islet endoplasmic reticulum and cytokine stress responses provides type 2 diabetes genetic insights。在本研究中,作者利用多组学分析揭示了胰岛细胞在应对环境压力时的转录反应,并将这些转录反应与非编码区变异关联起来,这项研究为理解T2D的发病机制提供了新的见解。

为了揭示环境应急对胰岛细胞的基因转录影响,作者首先使用来自非糖尿病供体的胰岛细胞,将其暴露在ER应激(thapsigargin)和促炎性细胞因子(IL-1b + IFN-g)中,作为研究胰岛应激反应的模型,然后通过RNA-seq分析胰岛细胞的基因表达变化。数据表明,约30%的基因(5131个基因)对ER应激或促炎细胞因子应激产生响应,这些基因中有85%单独对某个应激条件发生反应。其中ER应激激活了与未折叠蛋白反应(UPR)和ER蛋白处理通路相关的基因,如ATF4、ERN1/IRE1a、DDIT3/CHOP等,这些基因与β细胞功能和存活密切相关。另外,ER应激还显著诱导了胆固醇合成、氨基酸生物合成等代谢通路,表明ER应激对细胞功能的影响比单纯的蛋白质折叠更为广泛。与促炎性细胞因子相关的基因主要涉及细胞因子和趋化因子信号传导途径(如NF-kB通路),这些通路与胰岛β细胞功能障碍有关。促炎性应激还激活了多个固有免疫途径(如NLRP3炎症小体和RIG-I样受体信号通路),这些通路与早期1型糖尿病(T1D)的自身免疫激活有关。
为了进一步解读胰岛中不同细胞类型(尤其是α细胞和β细胞)对ER应激和促炎性细胞因子的反应,作者继续利用同样的方法处理非糖尿病供体的胰岛细胞,随后进行scRNA-seq分析。研究表明,β细胞表现出比α细胞更强的对ER应激和促炎性细胞因子的敏感性,β细胞对ER应激的反应中有98个基因被激活,而α细胞仅有35个,其中30个基因是两者共有的。在应激处理的胰岛细胞中,DDIT3、S100A6和MT1F等基因显著在β细胞中诱导表达,而α细胞中没有明显变化。β细胞在ER应激下可分为两个不同的亚群:β簇1(BC1):占94%的β细胞,显示出较为常规的应激响应;β簇2(BC2):占6%的β细胞,表现出更为强烈的与细胞死亡相关的基因表达,如蛋白酶体超家族基因PSMB8、PSMB9、PSMB10,这些基因与蛋白质降解和凋亡密切相关。这表明β细胞在T2D的病理机制中可能起着更重要的作用。
接下来,作者利用ATAC-seq绘制胰岛细胞在ER应激和细胞因子应激条件下的CRE的可及性图谱,旨在找出与这些应激反应相关的CREs,并解析它们如何调控基因表达。研究发现,大约14%的CREs(14,968个CREs)在应激处理后发生了显著变化,大多数应激响应的CREs是特异性的,仅有393个CREs在两种应激条件下共同响应。对这些应激响应CREs进行基因组注释发现,约91%的应激响应CREs位于距离最近的基因转录起始位点(TSS)1 kb之外,表明这些CREs可能是增强子,调控基因在应激条件下的表达。通过将这些CREs与胰岛细胞中表达的基因联系起来,研究发现应激响应的CRE的开放或关闭与相应基因表达的变化一致。转录因子结合位点的富集分析显示,ATF4、CHOP和NFIL3等UPR介导因子在ER应激条件下显著富集,而IRF8、IRF3以及NF-kB家族成员p65(RELA)的结合位点在促炎细胞因子应激下富集,表明不同的应激通过不同的转录因子调控其特定的基因表达。此外,这些应激反应不仅影响胰岛细胞的染色质结构和基因表达,还可能通过下调关键转录因子如PDX1和MAFA,削弱胰岛细胞的功能,进而导致葡萄糖代谢失调,促进T2D的发生。
作者还分析了与糖尿病相关的遗传变异和胰岛细胞在ER应激或促炎细胞因子应激条件下的CRE变化之间的关系。通过分析多个GWASs数据集,作者共发现161个与T2D、1型糖尿病或相关血糖性状相关的变异与应急反应 CRE的开放或关闭有关。其中,21个T2D相关变异与ER应激特异性开放的CRE重叠,24个T2D相关变异与细胞因子应激特异性开放的CRE重叠。例如,rs4744423通过增强FOSB/JUNB的结合来调控ER应激下AOPEP基因的表达,而rs6444081则通过破坏NRF2的结合位点,减少ETV5基因的表达,削弱胰岛功能。
文章的最后,作者选择了一个T2D相关的遗传变异rs6917676进行细致的研究。结果表明,rs6917676-T风险等位基因通过增强ER应激反应,增加MAP3K5表达,促进胰岛β细胞凋亡的分子机制。通过药物抑制MAP3K5,可能为减少T2D患者胰岛细胞凋亡、保护β细胞功能提供新的治疗策略。这些发现表明,特定的遗传变异可能通过改变胰岛细胞的应激反应,从而影响糖尿病的发生和发展。
模式图(Credit: Cell Metabolism)
总的来说,作者通过多组学研究剖析了ER应激和促炎症细胞因子应激对人类胰岛细胞转录调控网络的全基因组影响,并激发了将rs6917676风险等位基因与增强的ER应激反应性MAP3K5介导的β细胞凋亡联系起来的变异到功能研究。这项研究将遗传突变和环境因素的变化与胰岛细胞的转录影响关联起来,为理解T2D发病机制以及开发潜在的治疗方法提供了重要的借鉴意义。
参考文献
1. Halban, P.A., Polonsky, K.S., Bowden, D.W., Hawkins, M.A., Ling, C., Mather, K.J., Powers, A.C., Rhodes, C.J., Sussel, L., and Weir, G.C. (2014). b cell failure in type 2 diabetes: postulated mechanisms and prospects for prevention and treatment. Diabetes Care 37, 1751–1758.
2. Lawlor, N., Khetan, S., Ucar, D., and Stitzel, M.L. (2017). Genomics of islet (Dys)function and type 2 diabetes. Trends Genet. 33, 244–255.
3. Ashcroft, F.M., and Rorsman, P. (2012). Diabetes mellitus and the b cell: the last ten years. Cell 148, 1160–1171.
4. Suzuki, K., Hatzikotoulas, K., Southam, L., Taylor, H.J., Yin, X., Lorenz, K.M., Mandla, R., Huerta-Chagoya, A., Rayner, N.W., Bocher, O., et al. (2023). Multi-ancestry genome-wide study in >2.5 million individuals reveals heterogeneity in mechanistic pathways of type 2 diabetes and complications. Preprint at medRxiv.
5. Vujkovic, M., Keaton, J.M., Lynch, J.A., Miller, D.R., Zhou, J., Tcheandjieu, C., Huffman, J.E., Assimes, T.L., Lorenz, K., Zhu, X., et al. (2020). Discovery of 318 new risk loci for type 2 diabetes and related vascular outcomes among 1.4 million participants in a multi-ancestry meta-analysis. Nat. Genet. 52, 680–691.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


